티스토리 뷰
피마 인디언 당뇨병 데이터 세트를 이용해 당뇨병 여부를 판단하는 분류 모델을 학습시킨 뒤 지금까지 공부한 분류의 평가 지표들로 모델을 평가해봅시다.
피마 인디언 당뇨병 데이터 세트는 북아메리카 피마 지역의 원주민 Type-2 당뇨병 데이터 결과입니다.
데이터 세트는 아래의 피처로 구성돼 있습니다.
Pregnancies(임신 횟수), Glucose(포도당 부하 검사 수치), BloddPressure(혈압), SkinThickness(삼두근 뒤 피하지방량), Insulin(혈청 인슐린), BMI(체질량지수), DiabetesPedigreeFunction(당뇨 내력 가중치), Age(나이), Outcome(당뇨 여부, 0/1).
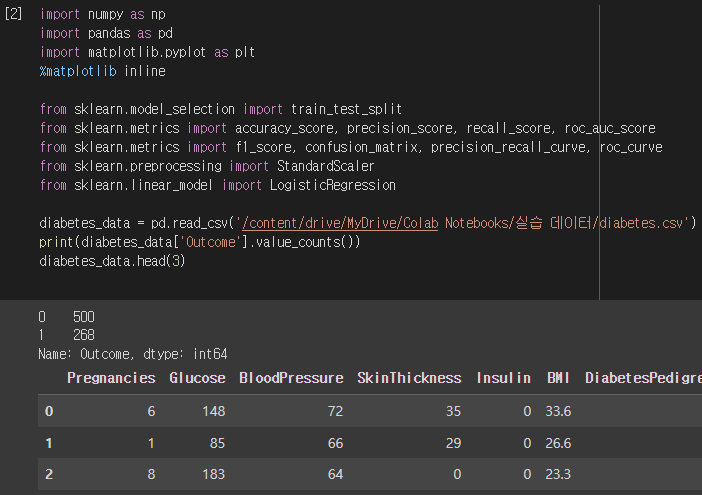
전체 데이터 중 Negative(0)이 500개로 Positive(1)보다 상대적으로 많습니다.
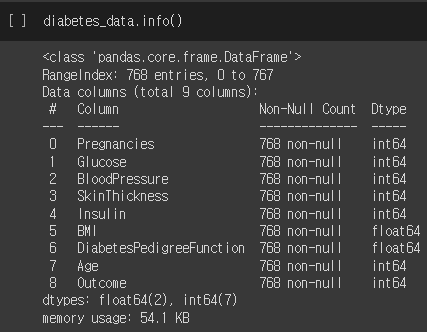
피처의 타입과 결손값의 개수를 확인합시다. 결손값은 없으며, 피처 타입이 모두 숫자형입니다.

로지스틱 회귀 분류 모델로 일단 학습을 진행했을 때, 재현율이 0.59정도로 꽤 낮은 것을 볼 수 있습니다. 태스크의 특성상 재현율을 높이는 것이 중요하므로, 임곗값 조정에 따른 정밀도/재현율 변화를 관찰하여 최적의 임곗값을 찾아봅시다.

임곗값이 약 0.4인 경우 정밀도와 재현율이 균형을 이루지만, 두 수치 모두 낮은 것을 볼 수 있습니다. 임곗값 조정을 통한 모델 성능 향상을 도모하기 전에, 데이터 자체에 대한 튜닝이 필요해보입니다.

df.describe() 메서드를 통해 각 피처의 분포도를 대략적으로 살펴보니, 값이 0이 될 수 없는 피처의 min 값이 0인 것을 볼 수 있습니다. Glucose, BlodePressure 등의 피처가 그러합니다. 이 표를 봐선 값이 0인 레코드가 몇 개인지는 파악할 수 없으므로, 피처별로 히스토그램을 그려 0인 레코드의 개수를 파악해볼 수 있습니다.

Glucose 피처의 경우 0인 레코드가 총 5개 입니다.

min이 0인 피처들에 대해, 0인 레코드가 각 피처에서 얼마만큼의 비율을 차지하는 지를 계산했습니다.

각 피처의 0인 요소를 각 피처의 평균값으로 대체했습니다.

또한 로지스틱 회귀 알고리즘은 숫자 데이터에 스케일링을 적용하는 것이 좋습니다. 0인 데이터를 평균값으로 대체하고 다시 학습을 수행하니 정밀도와 재현율이 상승한 것을 볼 수 있습니다.

0인 값 대체 및 피처 스케일링을 진행한 데이터 세트로 학습한 모델에 대해 최적의 임곗값을 다시 찾아봅시다. 위의 코드를 수행했을 때, 임곗값이 0.48일 때 F1 스코어가 가장 높다는 결과가 나왔습니다

임곗값 변화에 따른 정밀도/재현율 변화는 위와 같습니다.
'파이썬 머신러닝 완벽 가이드' 카테고리의 다른 글
| 파이썬 머신러닝 완벽 가이드 : 결정 트리 실습 - 사용자 행동 인식 데이터 세트 (1) | 2023.05.01 |
|---|---|
| 파이썬 머신러닝 완벽 가이드 : 분류(Classfication)와 결정 트리 (0) | 2023.05.01 |
| 파이썬 머신러닝 완벽 가이드 : 분류 모델의 평가 지표 (0) | 2023.04.30 |
| 파이썬 머신러닝 완벽 가이드 : 사이킷런으로 해보는 타이타닉 생존자 예측 (0) | 2023.04.29 |
| 파이썬 머신러닝 완벽 가이드 : 데이터 전처리 (0) | 2023.04.29 |
